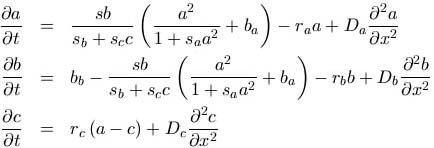Komplexe Muster durch konkurrierende Antagonisten
Muster, die punkten
Aus Sicht der Modellierung sind Streifenmuster simpel, da Oszillationen bzw. zeitliche Konstanz mathematisch
recht einfach zu verstehen und schließlich umszusetzen sind. Bei den ebenso simpel anmutenden Punkten sieht die Sache
schon anders aus. Man kann allerdings Flecken (Punkte oder kleine Streifen) auch aus Überlagerungen von
Streifenmustern gewinnen. Und mit Streifen haben wir ja bereits Erfahrungen gesammelt ...
Modellierung
Dem Aktivator-Substrat-Modell kann man hier bei anderen Simulationen begegnen:
Einfach beim Wellenmuster und gar doppelt beim Dreiecksmuster
von Conus marmoreus. Auch Aktivator-Inhibitor-Modelle sollten nicht unbekannt sein: Bei
den Streifenmustern waren sie zum Einsatz gekommen (hier und
hier).
Mit beiden Ansätzen lassen sich Streifen senkrecht wie parallel zur Wachstumskante erzielen. Daher ist es schon
vom Prinzip her interessant, was passiert, wenn zwei Antagonisten gleichzeitig eingesetzt werden,
wobei diese dann so eingestellt sind,
dass Tendenzen zum Streifenmuster senkrecht zur Wachstumskante mit Tendenzen zur Oszillation in Konkurrenz treten.
Einem Aktivator-Substrat-Modell wird daher ein zweiter Antagonist in Form eines Inhibitors, wie man ihn vom
Aktivator-Inhibitor-Modell her kennt, hinzugefügt. Wir sprechen dann vom Erweiterten Aktivator-Substrat-Modell.
Die mathematische Formulierung
Das System partieller Differentialgleichungen für das Erweiterte Aktivator-Substrat-Modell lautet wie folgt:
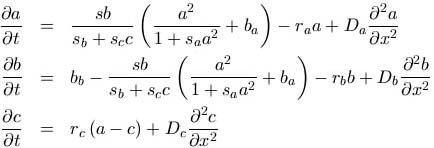
Man erkennt den typischen Zusammenhang zwischen Aktivator und Substrat, wo ein Zuwachs an Aktivator die gleiche
Menge an Substrat kostet:
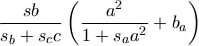
Offensichtlich geht hier ein Sättigungseffekt
bezüglich des Substrates ein, wobei die Wirkung des Inhibitors auf den Aktivator
in diesen Sättigungsterm mit einbezogen wurde.
Die Gleichung für den Inhibitor ist die bekannte, jedoch wird keine Grundproduktion berücksichtigt. Zu beachten ist,
dass die Wachstumsrate des Inhibitors dem Wert seiner Zerfallsrate angepasst ist. Wenn im folgenden Abschnitt also
von der Zerfallsrate des Inhibitors die Rede ist (so wird der Wert im Parameterfenster zu den Simulationen
automatisch benannt), so sollte man im Hinterkopf haben, dass es sich zugleich um die
- von der Aktivatormenge abhängige - Wachstumsrate handelt.
Mustersimulation und Analyse
Zum Erweiterten Aktivator-Substrat-Modell gibt es zwei Simulationen. Die Grundeinstellung für die erste der beiden sieht so aus:
|
Grundproduktion |
Zerfallsrate |
Diffusionskonstante |
Sättigungskoeffizient |
| Aktivator |
0.01 |
0.08 |
0.01 |
0.1 |
| Substrat |
0.08 |
0 |
0.3 |
0 |
| Inhibitor |
|
0.01 |
0.002 |
1 |
Das Muster zu diesen Parametereinstellungen kann sicher nicht einfach genannt werden. Wie kommt es zu Stande?
Schaltet man den Inhibitor aus (Wachstums- / Zerfallsrate rc = 0),
so verbleibt ein Aktivator-Substrat-Modell, das Streifen senkrecht zur Wachstumskante produziert.
Um die Wirkung des Inhibitors auf den Aktivator zu testen, kann man
leider nicht genauso vorgehen und das Substrat ausschalten (siehe Gleichung für Aktivator a).
Stärkt man aber die Wirkung des Inhibitors c durch die Einstellungen
|
Zerfallsrate |
Diffusionskonstante |
Sättigungskoeffizient |
| Inhibitor |
0.05 |
0.01 |
2 |
so oszilliert das Muster: Die komplexen Muster, die das Erweiterte Aktivator-Substrat-Modell produziert,
entstehen aus einem Ringen der Tendenzen zu parallelen und senkrechten Streifen.
Zurück zur Grundeinstellung: Hier ist die Inhibitorkonzentration so klein, dass eine hohe Substratkonzentration
zu ständiger Aktivatorproduktion führt. Senkt man die Grundproduktion für das Substrat ab, setzt die ständige
Aktivatorproduktion aus. Es entstehen Wanderwellen. Bei einer noch niedrigeren Grundproduktion
lösen sich die Linien mehr und mehr auf. Ein Fleckenmuster entsteht
(Grundproduktion bb = 0.02).
Schauen wir uns nun die zweite Simulation zum Erweiterten Aktivator-Substrat-Modell an.
Die Zweitversion zum Modell war für die hier gewählten Einstellungen nötig, weil sie sich teils sehr von den
Werten zu den bereits gesehenen Mustern unterscheiden.
|
Grundproduktion |
Zerfallsrate |
Diffusionskonstante |
Sättigungskoeffizient |
| Aktivator |
0.005 |
0.05 |
0.01 |
1 |
| Substrat |
0.03 |
0 |
0 |
1 |
| Inhibitor |
|
0.02 |
0.3 |
2 |
Wieder bekommt man ein komplexes Muster zu sehen. Schaut man sich das Muster jedoch in der Schwellenwert-Version mit einem Schwellenwert von 0.3 oder 0.4 an, sehen wir ein Fleckenmuster.
[Natürlicher erscheint es mit den Umstellungen xr = 200,
t1 = 5000, Dehnung um Faktor 1 in x-Richtung, weiterzeichnen nach
jeweils 20 Rechenschritten, Musterfarbe Schwarz (RGB-Wert 0,0,0) und schließlich Schwellenwert 0.2.]
Schaltet man wie bei der ersten Simulation in der Grundeinstellung durch Nullsetzen der
Zerfalls- bzw. Wachstumsrate (rc = 0)
den Inhibitor aus, erkennt man Oszillationen. Auch hier entstehen die komplexeren Muster aus einem Ringen
der Tendenzen zu Oszillation (Streifen parallel zur Wachstumskante) einerseits und zeitlicher Konstanz
(Streifen senkrecht zur Wachstumskante) andererseits. Es lässt sich also vermuten, dass entsprechend eine
Stärkung der Wirkung des Inhibitors gegenüber dem Substrat - äquivalent zu den Untersuchungen bei der ersten Simulation -
zu einem senkrechten Streifenmuster tendiert. Mit den einstellbaren Parameterwerten ist dies leider nicht
so klar machbar.
Stellt man die Wachstums- / Zerfallsrate für den Inhibitor auf 0.08, den Schwellenwert auf 0.6 ein und beobachtet
mittels der zugehörigen Visualisierung, was bei einem
Absenken seiner Diffusionskonstante (langsamere Verbreitung = lokal stärkere Wirkung) passiert,
kann man den Effekt des Inhibitors jedoch recht gut beobachten. Für Diffusionskonstante 0.06 schließlich
sieht man sogar das Umschwenken auf das senkrechte Streifenmuster.
zur Einstiegsseite